Tổng quan về chỉ số đa gen (Polygenic Risk Score - PRS)
Advisor: @Dat Nguyen
Writer: @Do Minh Nguyet
Giới thiệu
Chỉ số đa gen (PRS – Polygenic Risk Score) đã được nghiên cứu và lưu hành trong cộng đồng khoa học trong nhiều năm. Tuy nhiên, phải đến năm 2018, điểm số này mới được chứng minh là có tiềm năng sử dụng trong các nghiên cứu lâm sàng. Một nghiên cứu của Amit V. Khera và các đồng nghiệp tại Cardiovascular Disease Initiative of the Broad Institute tại Cambridge, Massachusetts đã xác định những người có nguy cơ cao mắc 5 bệnh phổ biến chỉ bằng cách dựa trên dựa bộ gen của họ [1].
Nhóm nghiên cứu đã sử dụng dữ liệu liên kết trên toàn hệ gen (GWAS – Genome Wide Association Study) và phương pháp xác định (imputation method) để đánh giá hàng triệu biến thể di truyền phổ biến liên quan đến bệnh động mạch vành, rung nhĩ, tiểu đường loại 2, bệnh viêm ruột và ung thư vú. Đối với mỗi bệnh, họ đã áp dụng một thuật toán tính toán kết hợp thông tin từ tất cả các biến thể thành một con số (PRS), phản ánh tính nhạy cảm di truyền của một người đối với các bệnh này.
Khi nhóm nghiên cứu thử nghiệm yếu tố dự báo đa gen của họ đối với bệnh tim trên 290.000 người tham gia tại Ngân hàng Biobank của Anh, họ nhận thấy rằng 8% dân số có nguy cơ đau tim cao gấp ba lần bình thường. Khera, người cũng là bác sĩ tim mạch và nhà di truyền học tại Bệnh viện Đa khoa Massachusetts cho biết: “Chúng tôi không thể tìm thấy chúng nếu không thực hiện xét nghiệm di truyền”. Sử dụng cách tiếp cận tương tự đối với bệnh viêm ruột và ung thư vú, nhóm nghiên cứu nhận thấy rằng chỉ số PRS của họ luôn có thể xác định một nhóm (từ 2-10% dân số) có nguy cơ đặc biệt cao. Kết quả của Khera cung cấp những bằng chứng thuyết phục ủng hộ việc sử dụng rộng rãi hơn các chỉ số PRS. kết qủa từ những nghiên cứu trên cho thấy được lợi ích của việc phát hiện sớm nguy cơ để tiến hành phòng bệnh. Một câu hỏi mới được đặt ra đó là làm cách nào để phát triển tiềm năng và nâng cao hiệu quả của việc sử dụng chỉ số đa gen trong các cơ sở y tế. Bài viết này khám phá sự phát triển của điểm sô đa gen và các phương pháp để giải quyết những thách thức xoay quanh việc sử dụng những chỉ số này.
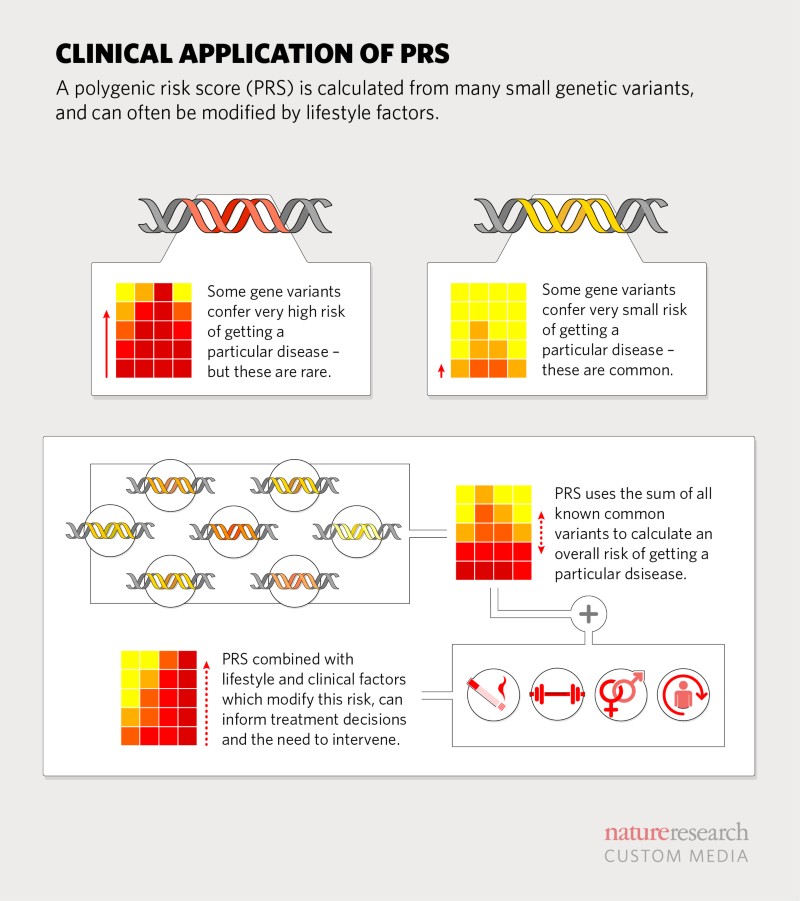
Từ một gen đơn lẻ đến các biến thể trên toàn bộ bộ gen
Xét nghiệm di truyền được sử dụng rộng rãi để chẩn đoán các bệnh đơn gen, chẳng hạn như bệnh xơ nang hoặc bệnh hồng cầu hình liềm, do đột biến ở một gen đơn lẻ. Các xét nghiệm này cũng có thể xác định những người mang gen liên quan đến bệnh tật không bị ảnh hưởng, cho phép họ đưa ra quyết định kế hoạch hóa gia đình sáng suốt hơn hoặc trong những trường hợp như bệnh Huntington, để lập kế hoạch trong trường hợp họ phát triển tình trạng bệnh trong tương lai.
Tuy nhiên, ở các bệnh thông thường, tiểu đường loại 2 là một ví dụ và nhiều bệnh thoái hóa thần kinh, có xu hướng đa gen – bị ảnh hưởng bởi một số lượng lớn các biến thể di truyền nằm rải rác trong bộ gen, cũng như các yếu tố môi trường và lối sống. Các công nghệ gen mới cho phép các nhà nghiên cứu giải trình tự nhanh chóng và không tốn kém. Các bảng gen lớn, tất cả các gen mã hóa protein (exome) hoặc toàn bộ bộ gen (giải trình tự toàn bộ bộ gen, WGS), cung cấp một bản khảo sát đầy đủ về cấu tạo gen của một người. Trong gần hai thập kỷ, các nhà di truyền học đã so sánh các bộ gen, tìm kiếm sự khác biệt để có thể giải thích tại sao chỉ một số người phát triển một số bệnh cụ thể.
Một nghiên cứu liên kết toàn bộ bộ gen (GWAS) có thể xác định sự biến đổi như vậy. Những biến đổi này thường ở dạng đa hình nucleotide đơn (SNP). Thông qua GWAS, các nhà nghiên cứu đã tìm thấy một số lượng lớn các biến thể liên quan đến bệnh, mặc dù vậy sự đóng góp của từng biến thể này có ảnh hưởng không đáng kể. Vài năm sau đó, họ đã phát triển các công cụ để xác định chính xác sự tác động của tập hợp hàng triệu các biến thể di truyền gây bệnh [2].
Xác định tính nhạy cảm của một người đối với các bệnh cụ thể đem lại rất nhiều lợi ích. Ví dụ, phát hiện của Khera cho thấy rằng ở những phụ nữ có nguy cơ cao bị ung thư vú, bắt đầu chế độ sàng lọc sớm hơn có thể cải thiện kết quả. Khera nói: “Mục tiêu của chúng tôi là trao quyền cho mọi người vượt qua bất kỳ khuynh hướng gây bệnh nào có trong DNA của họ”. Ngay cả khi một số bệnh hiện không thể chữa khỏi, PRS có thể được sử dụng để kết hợp các loại thuốc trong thử nghiệm lâm sàng. Desikan giải thích: “Một thử nghiệm về một loại thuốc trị liệu miễn dịch có thể mang lại nhiều lợi nhuận hơn bằng cách tập trung vào những người có điểm số đa nguyên miễn dịch cao”.
Cải thiện chỉ số đa gen
Một mối quan tâm quan trọng xung quanh việc thực hiện PRS trên lâm sàng là cho đến nay, điểm số phần lớn được tính toán từ trình tự DNA của người Châu Âu [3]. Martin nói: “Tần suất và mức độ tương quan với bệnh tật của các biến thể di truyền phổ biến ở người Mỹ gốc Phi khác với người Mỹ gốc Âu – và điều này làm giảm độ chính xác của PRS”. Martin tham gia vào các dự án toàn cầu khác nhau nhằm xác định đặc điểm của sự biến đổi bộ gen trong các quần thể đa dạng và phát triển các phương pháp thống kê để phân tích dữ liệu đa sắc tộc và cải thiện độ chính xác của PRS. Một trong những dự án này là Di truyền thần kinh tâm thần ở các dân cư châu Phi (NeuroGap). Bà nói: “Những cuộc di cư ban đầu của loài người ra khỏi châu Phi đã kéo theo sự đa dạng di truyền sang châu Âu, Đông Á và cuối cùng là châu Mỹ. Thực hiện các nghiên cứu di truyền lớn ở các quần thể châu Phi sẽ nhanh chóng cải thiện độ chính xác của PRS cho tất cả các quần thể.”
Vì nhiều tình trạng sức khỏe liên quan đến các yếu tố môi trường, lối sống và tính nhạy cảm di truyền, việc kết hợp PRS với các yếu tố nguy cơ đã biết khác sẽ cải thiện hơn nữa dự đoán nguy cơ và hỗ trợ xác định các ngưỡng lâm sàng [4]. Theo Ali Torkamani, một nhà di truyền học tại Viện Nghiên cứu Scripps ở La Jolla, California, có đủ bằng chứng để hỗ trợ việc sử dụng PRS trong các quyết định xung quanh điều trị bằng statin. Đối với những bệnh nhân được xác định là có nguy cơ phát triển bệnh mạch vành trên lâm sàng trung bình (từ các yếu tố nguy cơ lâm sàng thường được đo lường như hút thuốc, huyết áp cao và mức cholesterol), việc bổ sung thông tin nguy cơ đa sinh có thể giúp bác sĩ đưa ra quyết định kê toa statin.
Sử dụng chỉ số đa gen
Torkamani và nhóm của ông đã phát triển MyGeneRank, một ứng dụng có thể tính toán PRS của một cá nhân đối với bệnh mạch vành từ dữ liệu di truyền 23andMe của họ. Dữ liệu sức khỏe được thu thập trên thiết bị di động với một loạt bảng câu hỏi. Mục tiêu của họ là hiểu cách mọi người phản ứng khi nhận được điểm số và theo dõi bất kỳ thay đổi nào trong các hành vi liên quan đến sức khỏe sau đó.
Được bổ sung kiến thức về rủi ro di truyền thực sự có thể khuyến khích việc áp dụng các thay đổi lối sống lành mạnh trên phạm vi rộng hơn. Nghiên cứu GeneRisk ở Phần Lan được trình bày tại hội nghị ASHG năm 2018 cho thấy rằng việc cung cấp thông tin nguy cơ bệnh tim mạch được cá nhân hóa, dựa trên sự kết hợp của dữ liệu nguy cơ truyền thống và PRS đã góp phần thúc đẩy các hành vi lành mạnh. Ngay cả những người tham gia có nguy cơ thấp hơn cũng có cảm hứng giảm cân, ngừng hút thuốc hoặc đi khám bác sĩ. Các sáng kiến tương tự ở Estonia, nơi chính phủ đang tài trợ cho một chương trình định kiểu gen cho hơn 10% dân số của đất nước, đang điều tra việc sử dụng PRS cho bệnh tiểu đường loại 2 [5]. Các cá nhân được cung cấp tùy chọn để tìm hiểu điểm số của họ và những người có nguy cơ cao nhất được khuyến khích thực hiện thay đổi lối sống, chẳng hạn như giảm lượng đường và tăng cường tập thể dục, để ngăn ngừa hoặc trì hoãn sự khởi phát của bệnh tiểu đường.
Tạm kết
Dựa trên bộ dữ liệu di truyền, PRS sẽ sớm có sẵn để hỗ trợ mọi người có cuộc sống khỏe mạnh hơn. Khera nói: “Điều tuyệt vời về DNA là nó ổn định trong suốt thời gian tồn tại. Bạn có thể hình dung một tương lai không xa, trong đó, với 50 đô la, bạn sẽ nhận được một phiếu báo cáo tính nhạy cảm di truyền xác định những bệnh bạn có thể có nguy cơ mắc phải ngay từ khi còn nhỏ, vì vậy bạn có thể thực hiện các bước để ngăn ngừa chúng.”
Nguồn tham khảo
Bài viết được phiên dịch và chỉnh sửa từ ![]() Polygenic risk: What’s the score? của Nature Portfolio.
Polygenic risk: What’s the score? của Nature Portfolio.
[1] Amit V. Khera et al. “Genome-wide polygenic scores for common diseasesidentify individuals with risk equivalent to monogenic mutations”. In:Na-ture Genetics(2018).doi:https://doi.org/10.1038/s41588- 018-0183-z.
[2] Julian R. Homburger et al. “Low coverage whole genome sequencing enablesaccurate assessment of common variants and calculation of genome-widepolygenic scores”. In:Genome Medicine(2019).doi:https://doi.org/10.1101/716977.
[3] Alicia R. Martin et al. “Clinical use of current polygenic risk scores mayexacerbate health disparities”. In:Nature Genetics(2019).doi:https://doi.org/10.1038/s41588-019-0379-x.
[4] Ali Torkamani, Nathan E. Wineinger, and Eric J. Topol. “The personaland clinical utility of polygenic risk scores”. In:Nature Reviews Genetics(2018).doi:https://doi.org/10.1038/s41576-018-0018-x.
[5] Kristi L ̈all MSc et al. “Personalized risk prediction for type 2 diabetes:the potential of genetic risk scores”. In:Genetics in Medicine(2017).doi:https://doi.org/10.1038/gim.2016.103.