Overview of some popular reference genome versions
Writer: @Anh-Vu Mai-Nguyen
Advisor: @Thanh Nguyen
One of the indispensable pillars to build a foundation Precision Medicine is a reference genome. Over a history of more than 13 years summarized in this article Bộ gen tham chiếu đã được xây dựng như thế nào?,
The process of perfecting the human reference genome has achieved certain achievements. With the continuous progress of science and technology, the reference genome is constantly updated in the direction of becoming more and more accurate, synthesizing more information to represent the most general human genome. But also because of this updating process, many different versions of the reference genome have been born. This may not affect some analyses or may cause large differences in analysis results in the field of bioinformatics. This article will contribute to opening up an overview of the reference genome and the differences between popular versions.
“Shape” of the reference genome
Reference Genome (English: Reference Genome or Reference Assembly) is a digital database of nucleic acid chain, was assembled (assembly) by scientists, considered as a genome of an ideal organism for a species. Because it is assembled from the DNA sequences of a group of experimental individuals, the reference gene does not completely represent the genes of any one individual. Instead, the reference gene provides a mosaic đơn bội of different DNA sequences from each experimental participant.
The simplest format of a reference genome is a file fasta contains sequences of nucleic acids or amino acids, divided into many “contigs” (usually chromosomes). However, this information is only sufficient for one type of analysis: alignment . Other file types – gene annotation files (genome annotation) GTF or GFF allow for more downstream analysis because they show gene properties such as transcriptional region coordinates, exons, introns, etc.
Some versions of the reference genome
Currently, most of the analyses and published articles in the field of human genetics or bioinformatics use two main versions: hg19 (also known as GRCh37 – Genome Reference Consortium Human Build 37, named after the 37th meeting of this conference) or hg38 (GRCh38). However, for each version, there are many additional versions, published by different research units.
hg19 appendices
GRCh37 (NCBI)
GRCh37, The full name is Genome Reference Consortium Human Build 37, named after the 37th meeting of the Genome Reference Consortium conference. The official version was built by Genome Reference Consortium, published on February 27, 2009. The file containing the official reference genome is released and maintained by NCBI. (National Center for Biotechnology Information).
In this appendix, the names of chromosomes 1 to 22 are named NC_00000a.b with a corresponding to 1 to 22 and b is the version number. Similarly, chromosomes X and Y have a=23 and a=24, respectively.
Currently, this appendix has been updated by NCBI to the 13th time, the file name is GCF_000001405.25_GRCh37.p13_genomic.fna.gz with MD5sum 46e212080d30b1a24abec3eab36dbacd.
Official source:
File containing reference genome FASTA
Folder containing related files GCF_000001405.25_GRCh37.p13
b37
The Broad Institute created a new reference genome based on NCBI’s GRCh37, named b37. Compared to the original, b37 has a few changes such as consecutive segment names, and low-confidence bases are converted to N characters according to IUPAC code.
In this appendix, the names of chromosomes 1 to 22 are numbered from 1 to 22, respectively. The X and Y chromosomes are X and Y, respectively.
Official source:
humanG1Kv37
This is the appendix used in the analysis of 1000 Genomes Project.
The humanG1Kv37 appendix is equivalent to version b37 but does not contain decoys for human gammaherpesvirus 4 (NC_007605).
In this appendix, chromosomes 1 to 22 are labeled 1 to 22, respectively. Chromosomes X and Y are labeled X and Y, respectively.
Official source:
File containing reference genome FASTA
Folder containing related files phase2_reference_assembly_sequence
hg19 (UCSC)
The University of California at Santa Cruz (UCSC) created an hg19 appendix based on GRCh37. The appendix has a reference genome file hg19.fa.gz with MD5sum: 806c02398f5ac5da8ffd6da2d1d5d1a9.
In this appendix, chromosomes 1 to 22 are named chr1 to chr22, respectively. Chromosomes X and Y are chrX and chrY, respectively.
Official source:
File containing reference genome FASTA
Folder containing related files Golden Path
hg38 appendices
GRCh38 (NCBI)
GRCh38, The full name is Genome Reference Consortium Human Build 38, named after the 38th meeting of the Genome Reference Consortium conference. The official version was built by Genome Reference Consortium,
published on February 28, 2019. The file containing the official reference genome is released and managed by NCBI (National Center for Biotechnology Information). Currently, this version has been updated by NCBI for the 13th time, the file name is GCA_000001405.28_GRCh38.p13_genomic.fna.gz with MD5sum: f28b7146e0f30efa58447eceb32620a3.
In this appendix, the names of chromosomes 1 to 22 are named CM000a.2 with a corresponding to 663 to 684. Similarly, chromosomes X and Y have a=685 and a=686, respectively.
Official source:
- Updates at this link. File containing the reference genome FASTA
Folder containing related files GCA_000001405.28_GRCh38.p13
GRCh38 Resource bundle (Broad Institute)
In addition to the reference genome, this appendix also includes standard databases for analyzing human genome sequencing data via GATK – Genome Analysis Toolkit.
The Broad Institute is currently contributing to the global standard for genetic analysis using GRCh38/hg38 through its standard database. The International Genome Sample Resource (IGSR) project also uses this dataset as a basis for analysis in Phase 3.
In this appendix, chromosomes 1 to 22 are labeled chr1 to chr22, respectively. Chromosomes X and Y are chrX and chrY, respectively.
Official source:
Detail comparison
In addition to the large change in coordinates, what makes hg38 more useful for analysis than hg19 is the large number of alternate regions. These alternate regions, named “*_alt”, represent sequences that are common in the human population but are quite different from the sequence in hg38. The presence of these alternate regions has made the analysis of different populations around the world more precise.
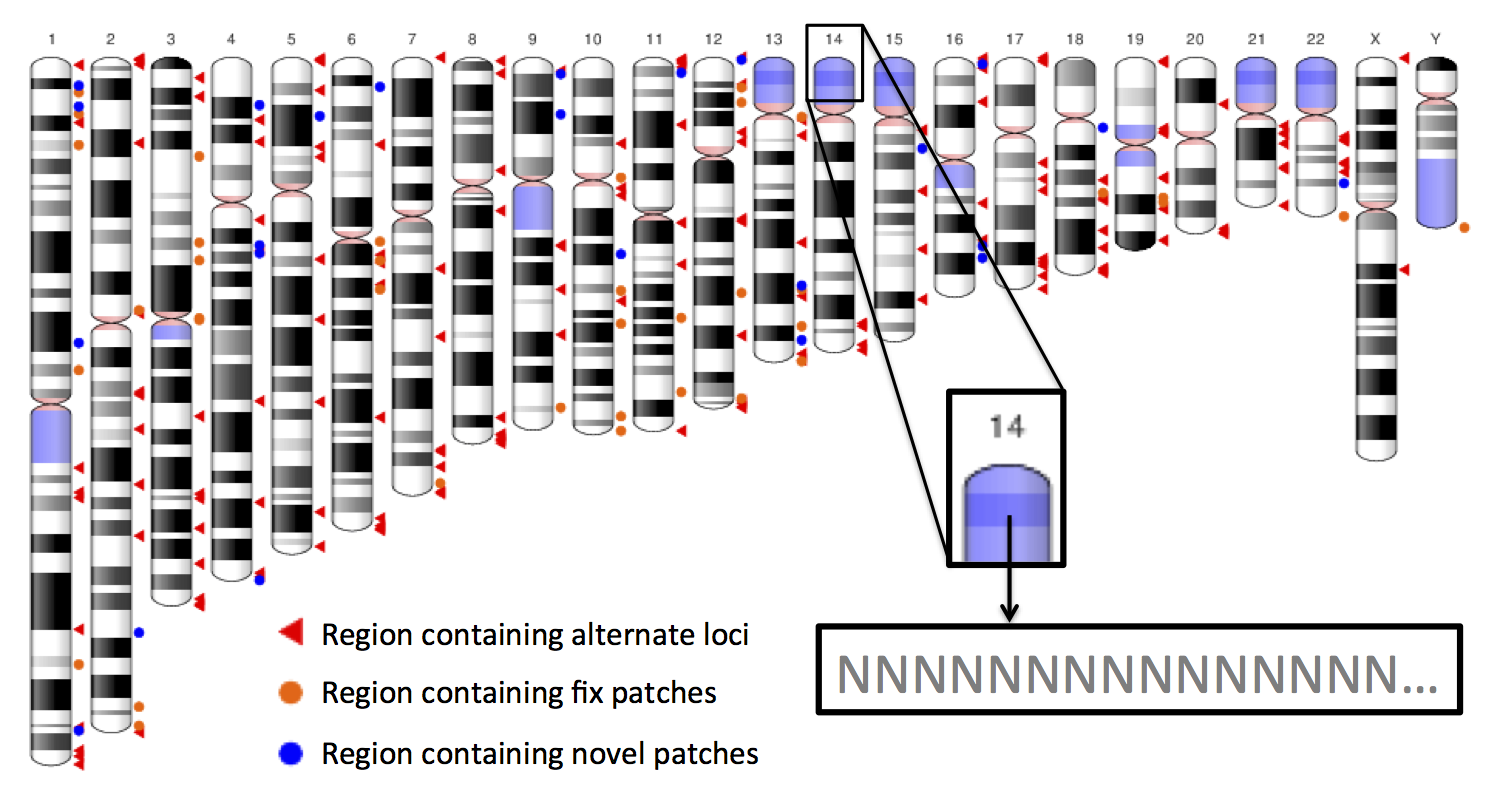
Ngoài ra, còn có sự khác biệt ở tên của các đoạn liên tiếp, biểu diễn vùng tâm động (centromere) và gen ti thể (mitochondrial genome).
Ở trong bài viết này, chúng tôi trực tiếp so sánh các đoạn liên tiếp giữa các phiên bản bộ gen tham chiếu thông qua tính giá trị tổng kiểm của mã hóa md5 (md5checksum). Trước tiên, chúng tôi đã tiến hành chuẩn hóa cách biểu diễn các trình tự trong các phụ bản và phiên bản khác nhau:
Chuyển toàn bộ chữ in thường thành in hoa (chữ in thường thể hiện vùng repetitive)
Chuyển toàn bộ kí tự khác A, T, G, C, N thành N. Lý do vì một số phiên bản có thể sử dụng kí hiệu của IUPAC.
Kết quả được miêu tả trong tệp sau:
Từ kết quả trên, chúng tôi rút ra một số quan sát:
Phụ bản humanG1Kv37 có thêm đoạn mồi tổng hợp có tên “hs37d5” so với phụ bản b37 của Broad Institute.
Cả 2 phiên bản GRCh37 và GRCh38 của NCBI đều không chứa chrEBV hay NC_007605 (gammaherpesvirus 4 ở người) so với các phụ bản khác. Ngoài ra, trong những “đoạn liên tiếp chính” (primary contigs), các phiên bản do NCBI phát hành này cũng khác hầu hết các phụ bản ở nhiễm sắc thể Y.
Phụ bản hg19 của UCSC khác các phụ bản khác của hg19 ở gen ti thể M, trong khi lại giống nhiễm sắc thể Y với GRCh37 của NCBI.
Ngoài ra trong phiên bản GRCh38, phụ bản của NCBI khác với Broad Institute ở nhiễm sắc thể số 5, số 14, số 19, số 21, số 22. Phân tich cụ thể cho thấy các khác biệt này chủ yếu nằm ở kí hiệu của chuỗi trên vùng tâm động.
Bình luận mở rộng
Một kết quả khá bất ngờ cho thấy mặc dù cùng phiên bản, nhưng mỗi phụ bản phát hành bởi một đơn vị nghiên cứu khác nhau lại không hoàn toàn trùng khớp. Điều này đặc biệt ảnh hưởng đến khả năng tái lập kết quả giữa các nghiên cứu khác nhau, nhất là với những biến dị hiếm gặp trên những vùng đặc biệt.
Thêm vào đó, mặc dù phiên bản mới hơn của bộ gen tham chiếu là GRCh38 đã được cập nhật từ năm 2013, nhưng cho đến nay vẫn rất nhiều phân tích sử dụng phiên bản cũ hơn là GRCh37 (phát hành năm 2009). Nguyên nhân chủ yếu của việc này do khá nhiều công cụ, bộ cơ sở dữ liệu vẫn chưa cập nhật, nên các phân tích vẫn phải sử dụng bộ gen tham chiếu cũ. Các phân tích sử dụng phiên bản khác nhau có sự khác biệt đáng kể về toạ độ, cũng như độ tin cậy của phân tích, điều này cũng gây ra không ít khó khăn cho việc thống nhất, chia sẻ kết quả. Điều này, bên cạnh lý do về công nghệ, cũng là một phần lý do khiến GRC vẫn chưa cập nhật phiên bản tiếp theo của hệ gen tham chiếu ở người.
Tại Việt Nam, từ năm 2018, khi bắt đầu những bước đầu tiên của dự án giải mã 1000 hệ gen người Việt, viện nghiên cứu dữ liệu lớn VinBigdata qua nghiên cứu kĩ lưỡng, đã tin tưởng và sử dụng bộ gen tham chiếu GRCh38 được đóng gói trong Resource bundle của Broad Institute. Qua đó, dự án có thể tận dụng được những bộ cơ sở dữ liệu và công cụ mới nhất, đồng thời đảm bảo được mức độ tin cậy cao với dữ liệu đầu ra.